1、安装上传FTP软件
为了获得稳定且易操作的数据上传体验,我们建议您事先在电脑上安装 FTP 上传软件。如果您熟悉使用命令行,或者您需要上传的文件够小足以在浏览器中完成上传,则可以跳过这一步骤。
云序生物推荐您使用 FileZilla 这款 FTP 软件。我们已经为您下载好了 FTP 软件 FileZilla 客户端的安装文件,请在您的实验报告文件夹的下述路径中找到与您系统版本(32位或64位 Windows 操作系统)对应的安装文件,然后按照指引完成软件安装:
Report\Sequence_Results\GEO_Upload\ftp software
如果您使用的电脑操作系统为 macOS 或 Linux,可以前往 FileZilla 官网的下载页面(https://filezilla-project.org/download.php?type=client)下载与您操作系统对应的客户端版本。
2、准备数据文件
云序生物所开展的实验项目中,需要上传到 SRA 的常见文件类型有:
(1)测序原始结果文件,例如 fasta、fastq 等格式的文件。
(2)测序结果与参考基因组比对结果文件,例如 bam 格式。
如需上传压缩文件,请使用 Linux 命令行 gzip/bzip2/tar 命令进行压缩(例如云序常见提供的 gz 格式的测序结果压缩文件),勿使用 Windows 下常见的压缩工具所生成的 zip、rar 等格式。对于测序原始数据,云序生物已经为您将 fastq 压缩为 gz 格式,您可以直接用于上传。如果您对压缩文件的格式不熟悉,也可以选择不压缩直接上传原文件。我们建议您在文件名中*使用英文字母、阿拉伯数字以及英文标点符号,勿使用其它类型的字符。
3、SRA上传
进入 SRA 数据库的上传页面(Submission Portal)
https://submit.ncbi.nlm.nih.gov/subs/sra/
点选“Log in”,进入账户登陆界面。由于 NCBI 政策调整,现*支持使用第三方账户登入,请选择一个您常用的第三方账户。在中国大陆网络环境下,建议您使用微软(Microsoft)账户登入。
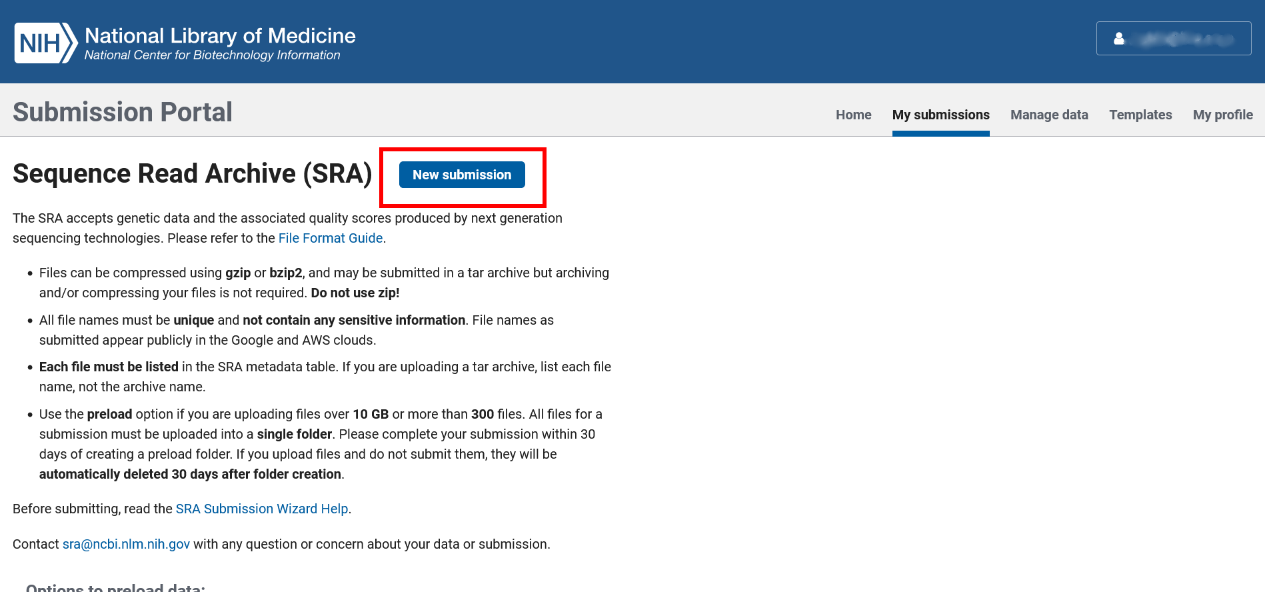
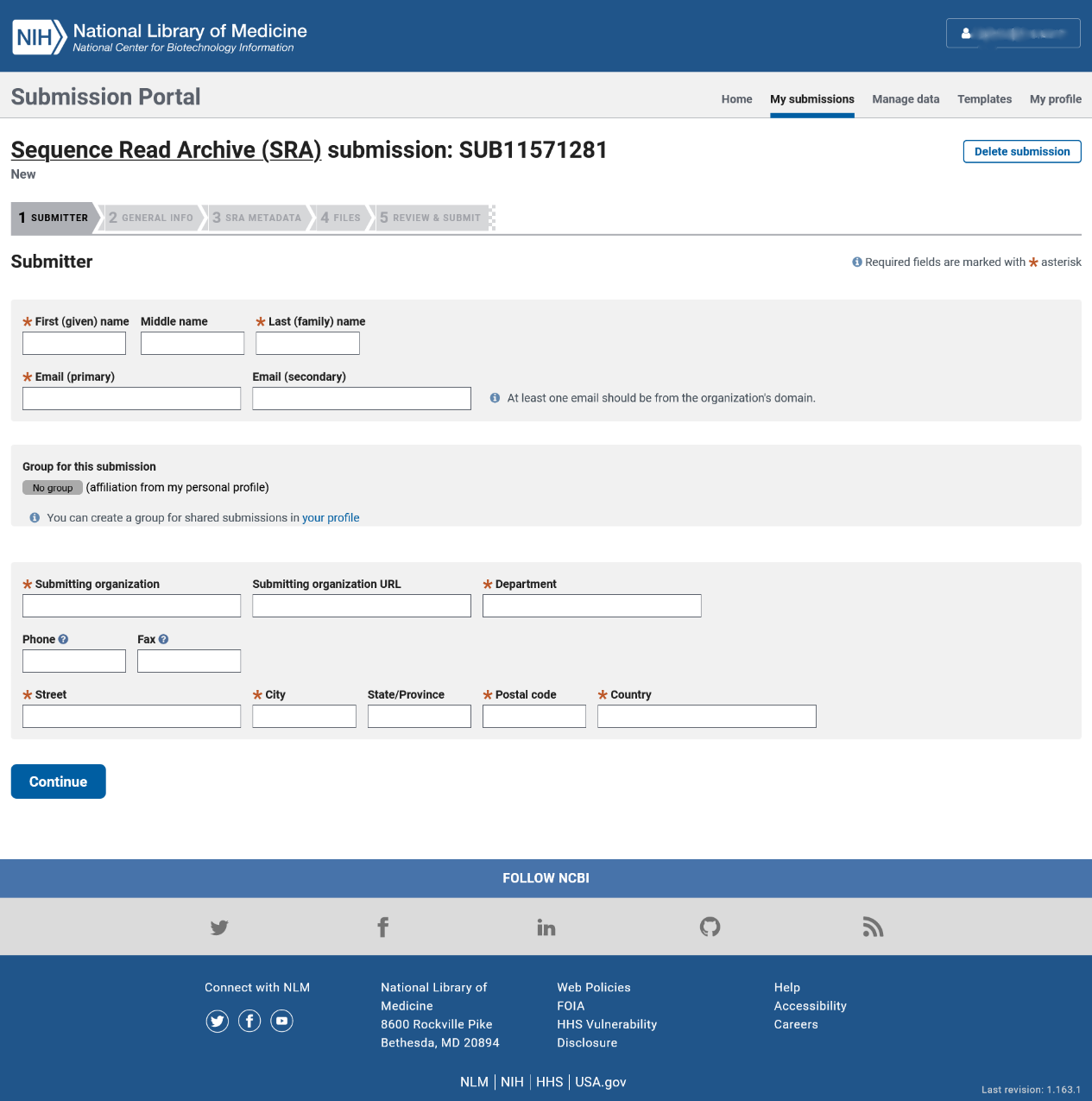
**步:上传者信息(SUBMITTER)
请至少填满所有带星号的空格。
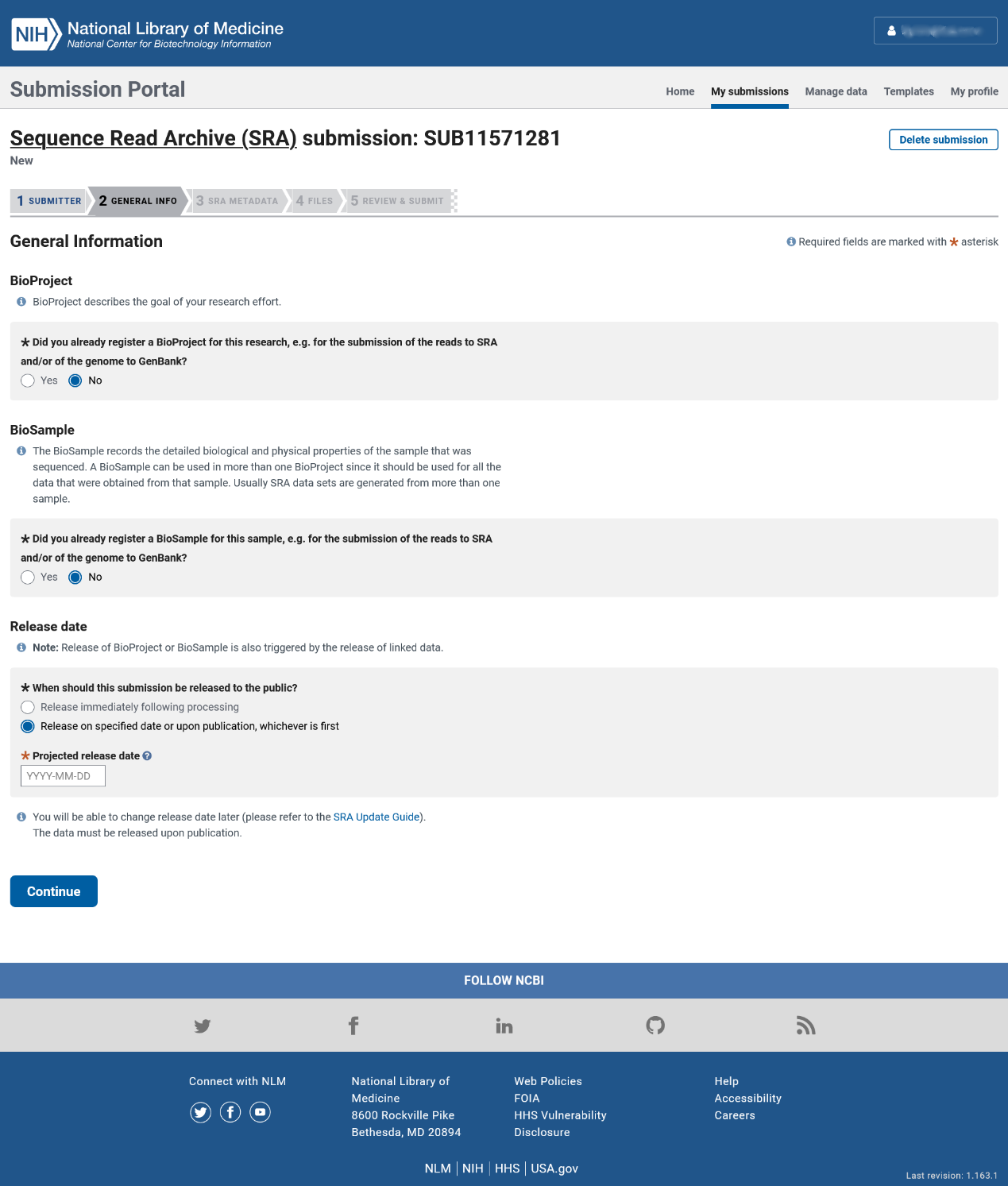
第二步:基本信息
如果您之前没有为此研究项目注册过 BioProject 和 BioSample 号,请如下图所示在前两个问题勾选“No”选项。在**一个问题中,您可以选择何时公开您上传的数据,我们建议您选择“Release on specified date or upon publication, whichever is first”选项,并指定一个您希望的日期。
第三步:项目信息
一般只需填写标题(Project Title)并描述实验目的(Public Description)即可,剩余部分一般可以留空。该部分信息*用于公开展示您对此项目的描述,无特殊格式要求。
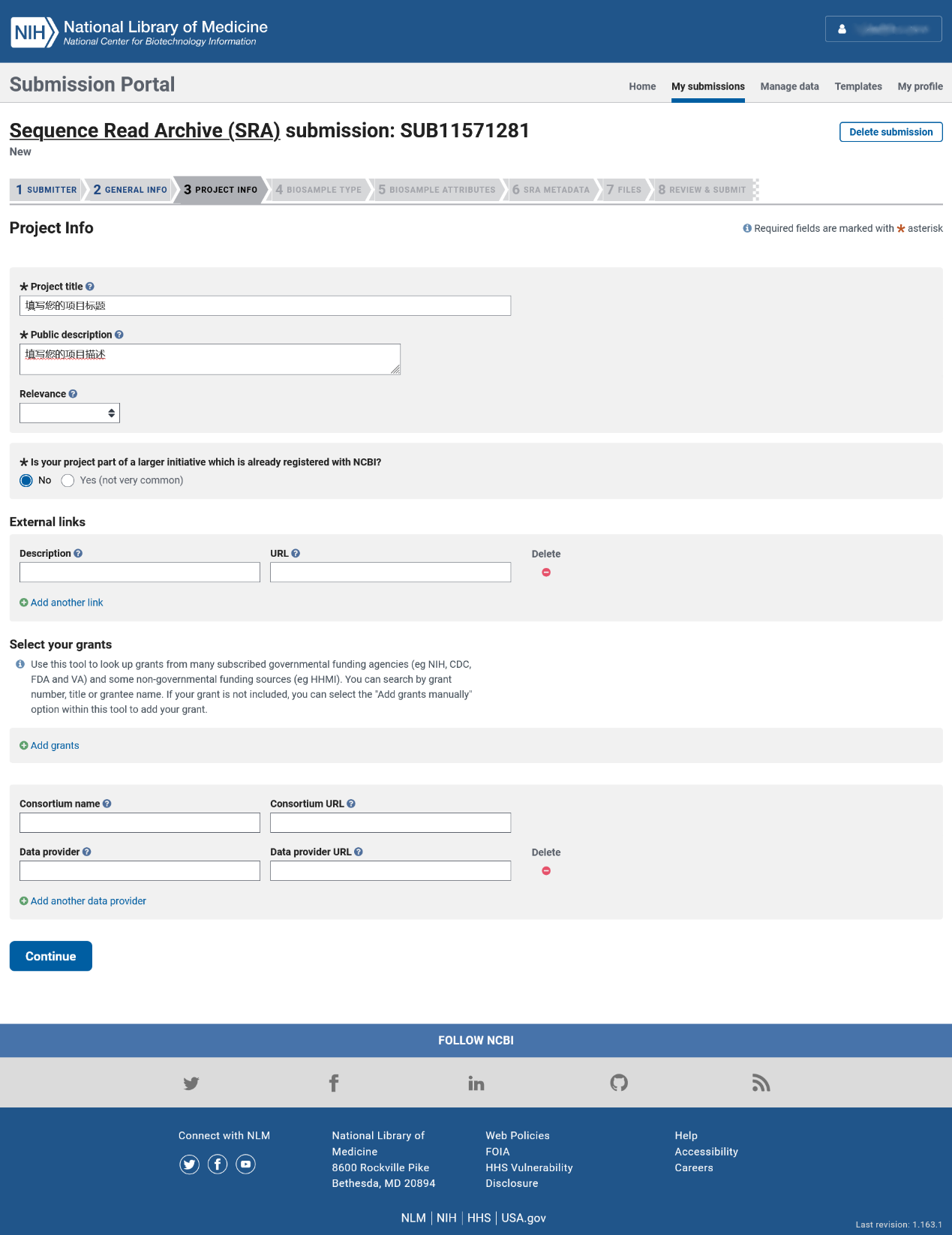
第四步:样本类型
对于大多数组织、细胞等常规类型的样品,请您使用默认的“All packages”标签栏,再输入框里输入您实验物种的拉丁名(例如:Homo sapiens),随后会在下方实时显示筛选过后的选项,请勾选符合描述的选项(例如:Human);如果您的样品是包含环境样品的宏基因组样品,请您选用“Packages for MAG submitters”和“Packages for metagenome submitters”这两个标签栏中合适的选项。
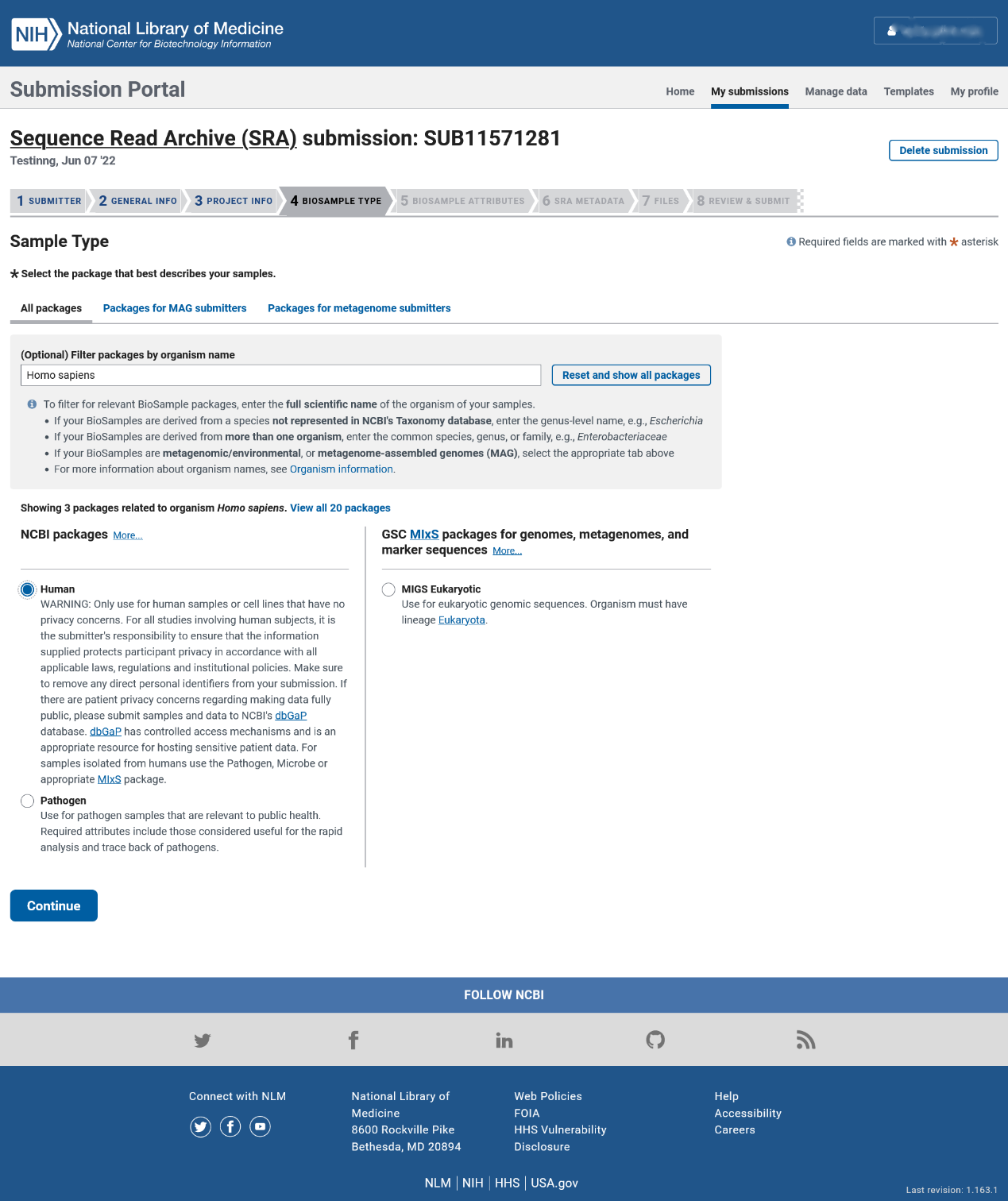
第五步:样本详情
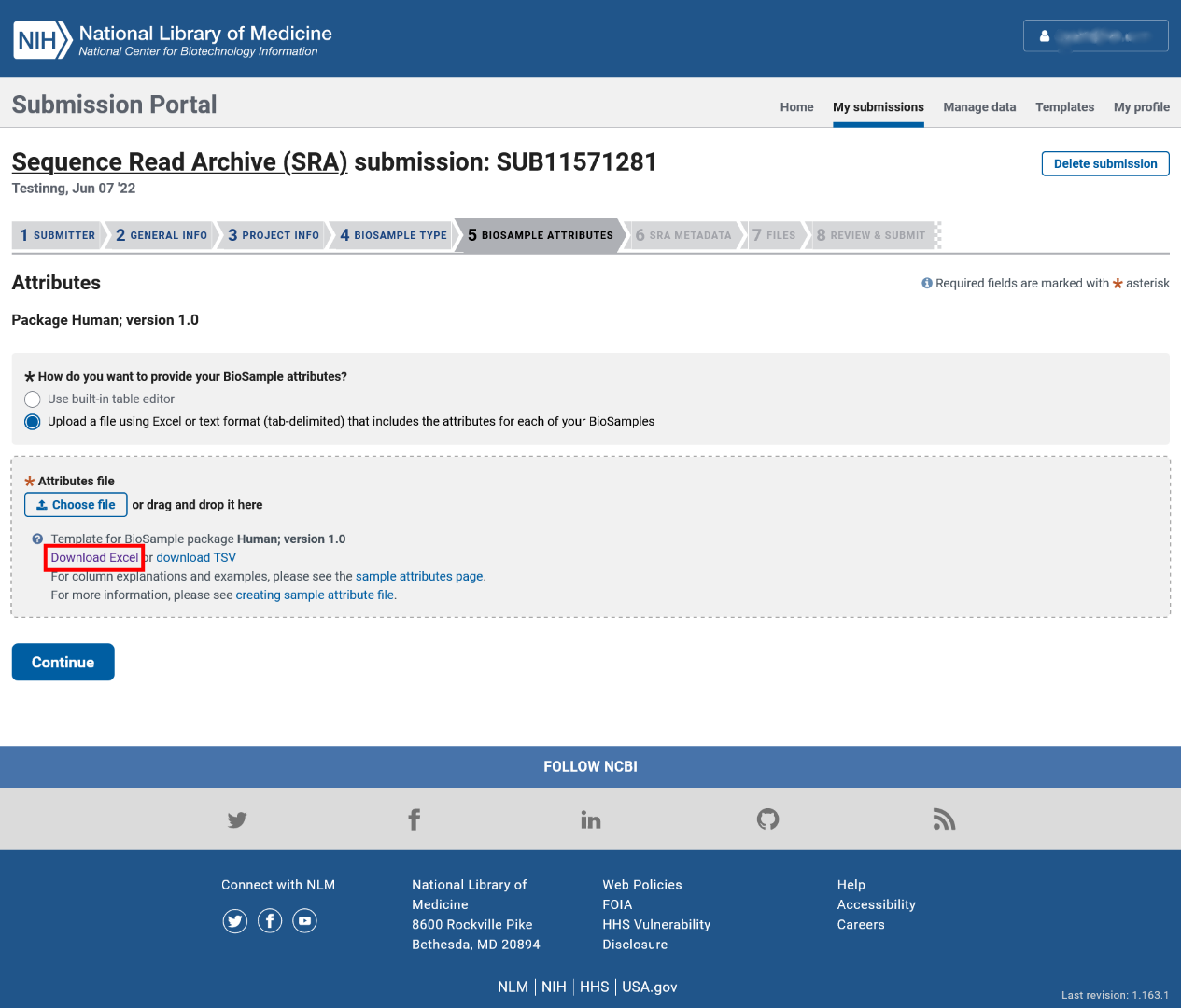
我们建议您勾选“Upload a file using Excel or text format (tab-delimited) that includes the attributes for each of your BioSamples”并点击下图红框内的“Download Excel”超链接下载模板表格文件,以便在本地电脑上慢慢填写表格,待填写完成后再点击“Choose File”按钮上传本地表格文件。
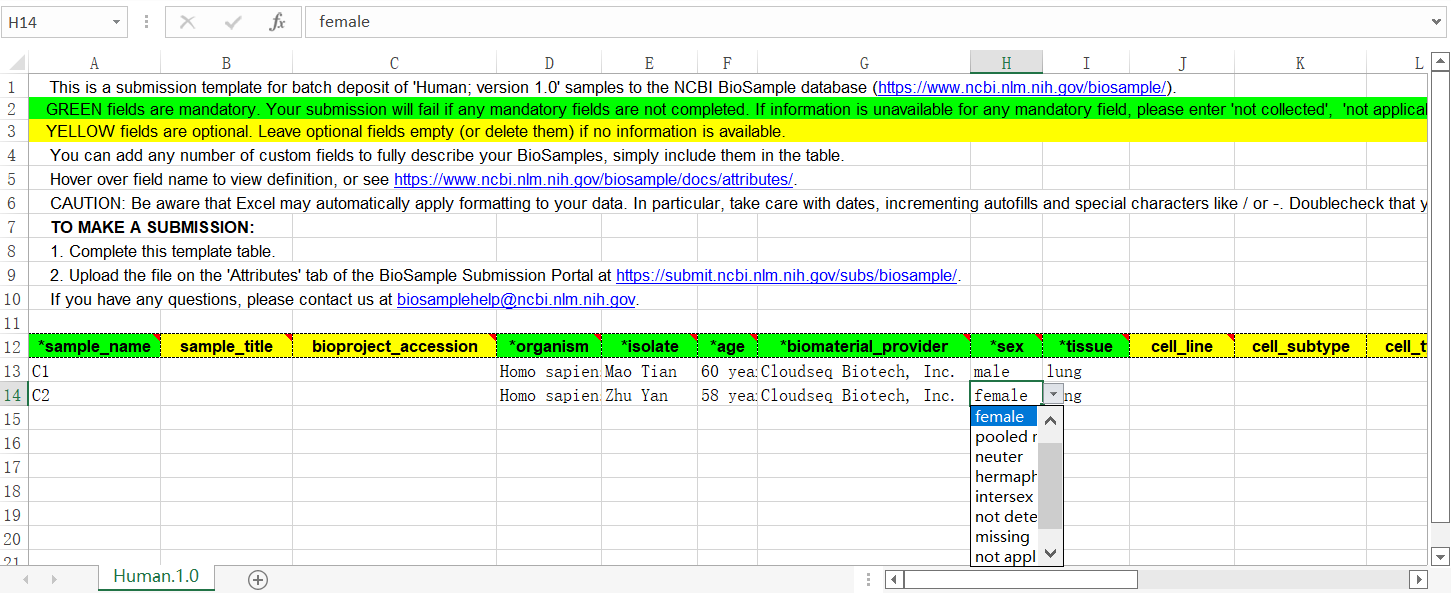
(1) 样品名称(sample name):无特定格式要求。
(2) 物种(organism):实验物种的拉丁名。
(3) 个体(isolate):为该实验样品个体取一个名字,无特定格式要求。
(4) 年龄(age):格式为【数字】+【时间单位】。
(5) 单位(biomaterial_provider):实验室或PI的名称和地址等信息,无特定格式要求。
(6) 性别(Sex):请从下拉菜单中选取符合条件的选项,而不要手动填写。
(7) 组织(tissue):样品来源的组织类型,无特定格式要求。
对于黄色单元格所在的纵列,您可以按需填写。如果您想补充更多信息以更好地描述您的样品,也可以手动添加纵列。
第六步:SRA 元数据
我们建议您勾选“Upload a file using Excel or text format (tab-delimited)”并点击下图红框内的“Download Excel spreadsheet”超链接下载模板表格文件,以便在本地电脑上慢慢填写表格。
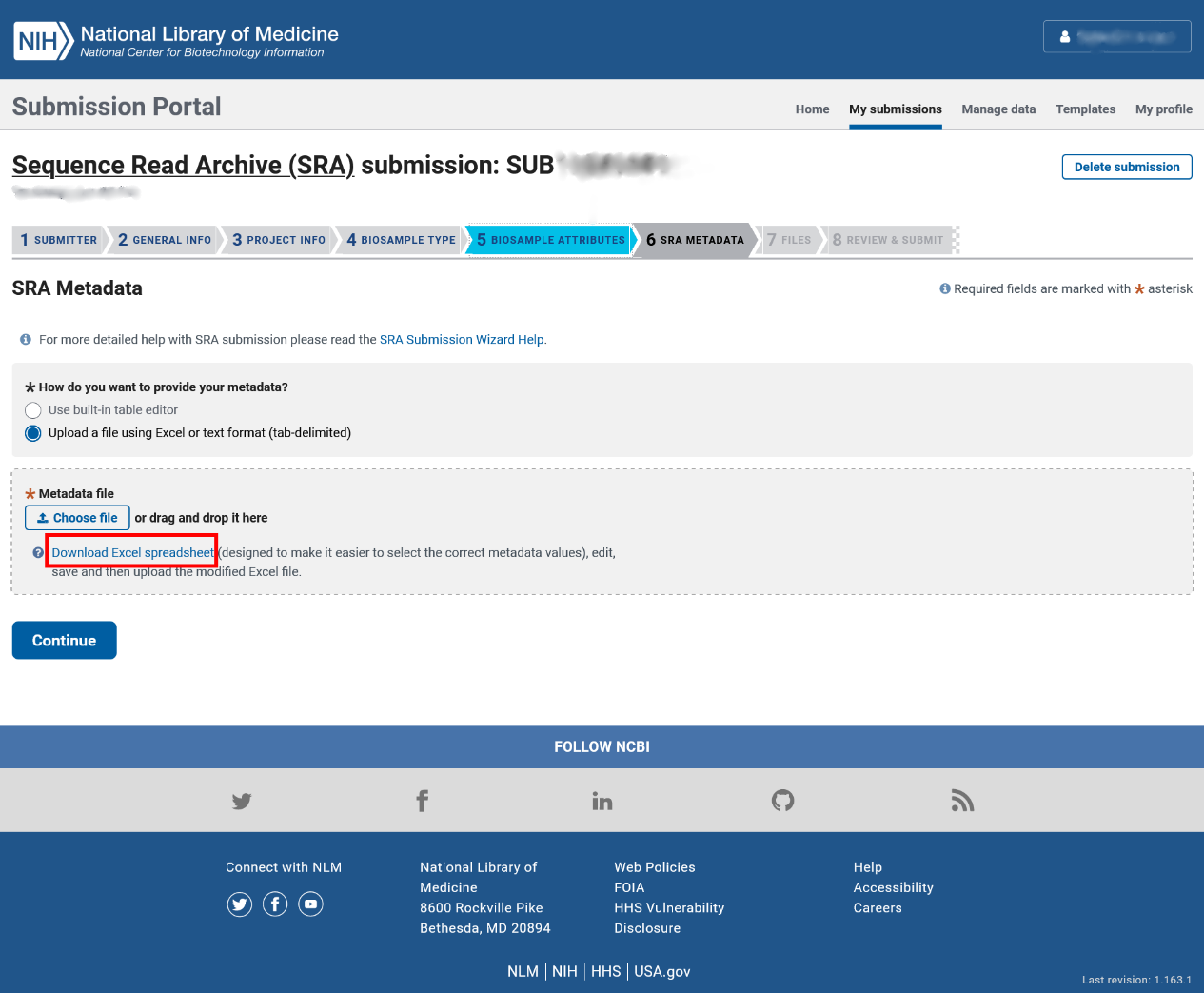

下滑查看内容
(1) 样品名称(sample name):无特定格式要求,但需与第五步中上传表格使用过的样品名称保持一致。
(2) 文库ID(library ID):无特定格式要求。
(3) 标题(title):建议采用 {methodology} of {organism}: {sample info} 这样的格式,例如“RNA-Seq of mus musculus: adult female spleen”。
(4) 文库方法(library_stratagy):请从下拉菜单中选取。点击标题超链接,您可以查看到各个选项的说明。例如,对于转录组的测序类项目,请选择 RNA-seq。
(5) 文库来源(library_source): 请从下拉菜单中选取。点击标题超链接,您可以查看到各个选项的说明。例如,对于转录组的测序类项目,请选择 TRANSCRIPTOMIC。
(6) 文库筛选(library_selection):请从下拉菜单中选取。点击标题超链接,您可以查看到各个选项的说明。例如,对于实验过程中去除了rRNA类的项目,请选择 Inverse rRNA。
(7) 文库端数(library_layout):请从下拉菜单选取单端测序(single)或双端测序(paired)。
(8) 测序平台(platform):请从下拉菜单中选取。点击标题超链接,您可以查看到各个选项的说明。例如,云序生物绝大多数的测序项目,都请选择 ILLUMINA。
(9) 测序仪器(instrument_model):请从下拉菜单中选取。点击标题超链接,您可以查看到各个选项的说明。您可以在云序生物提供的 HTML 报告中找到测序仪器的描述,例如 Illumina NovaSeq 6000。
(10) 方法描述(design_description):无特定格式要求。
(11) 文件类型(filetype):请从下拉菜单中选取。点击标题超链接,您可以查看到各个选项的说明。例如,云序生物绝大多数的测序项目,都会提供 fastq 格式文件。
(12) 文件名(filename):此行所描述的文件的名称(含文件后缀名)。如果存在多个文件,例如对于双端测序而言,请您将 R1 测序文件的文件名填写在“filename”一列(例如“C1.IP_R1.fastq.gz”),然后再将 R2 测序文件的文件名填写在“filename2一列(例如“C1.IP_R2fastq.gz”)。
(13) 参考基因组(assenbly 或 fasta_file):此行上传的数据是 bam 格式等与基因组比对后生成的文件,请提供参考基因组的信息。如果是 NCBI 上的公开基因组,请在 assembly 一列填写基因组版本名称;如果使用的是自定义的本地基因组文件,请在 fasta_file 一列填写基因组文件的名称。如果此行数据描述的文件不涉及基因组比对(alignment)操作,这两列都可以留空。
第七步:数据上传
将第六步填写的表格中所列出的文件全部上传至 SRA 的服务器,我们建议您使用 FTP 上传以获取稳定的数据上传体验。
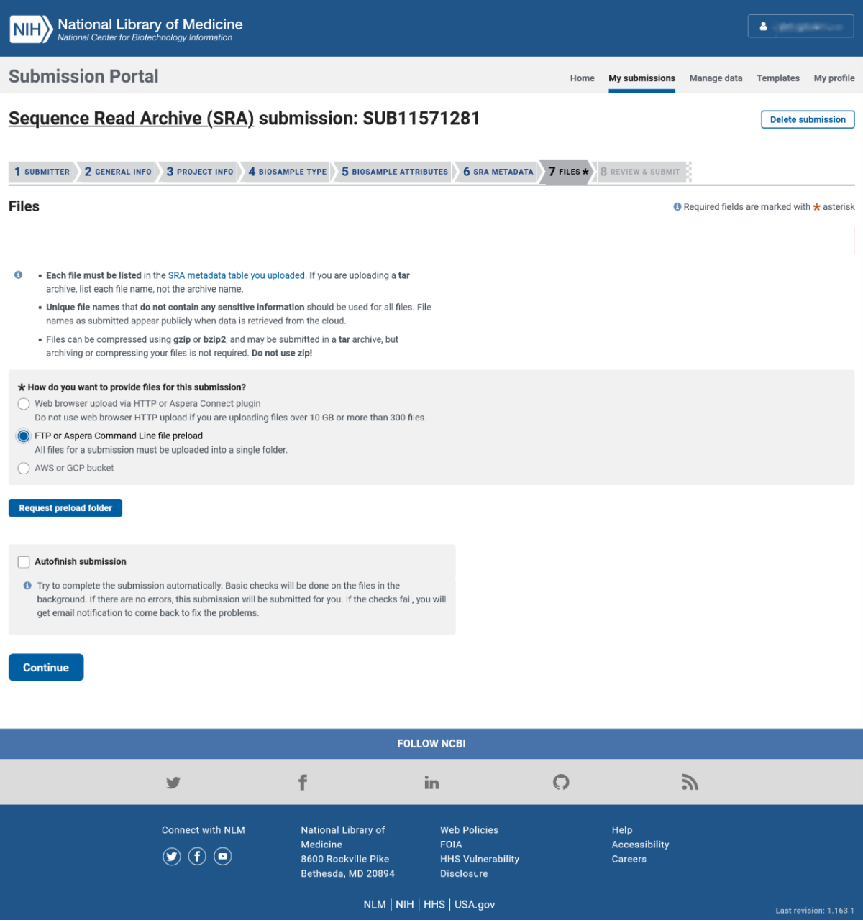
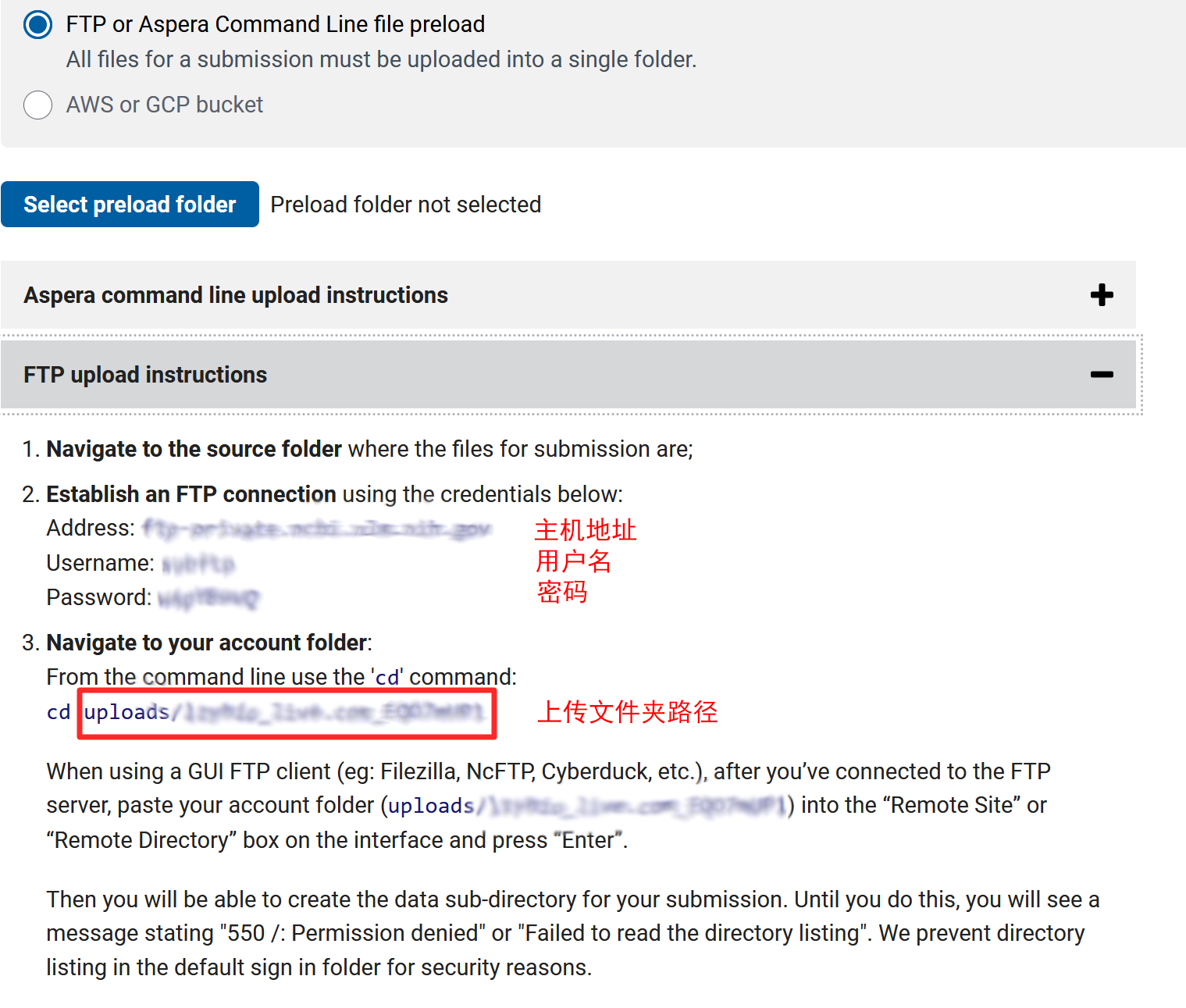
如果您的电脑上安装了安全软件或开启了防火墙,建议您暂时停用,或单独允许 FileZilla 软件的 FTP 连接。当 FTP 连接初次建立时,FileZilla 软件会报错“错误:读取目录列表失败”,系正常现象。您需要按照以下操作方法,手动填写远程站点的目录,软件才能正确读取目录:在如下图所示的“远程站点”位置中填入从上图中复制的“上传文件夹路径”(请复制粘贴整个字符串,前后不要留空格,不要加引号),按下回车键,就进入了您在 SRA服务器上的远程文件夹。随后,请您将本地电脑上需要上传的所有文件拖拽进入 FileZilla 软件如图所示的右下角的空白区域,软件将会开始自动上传。
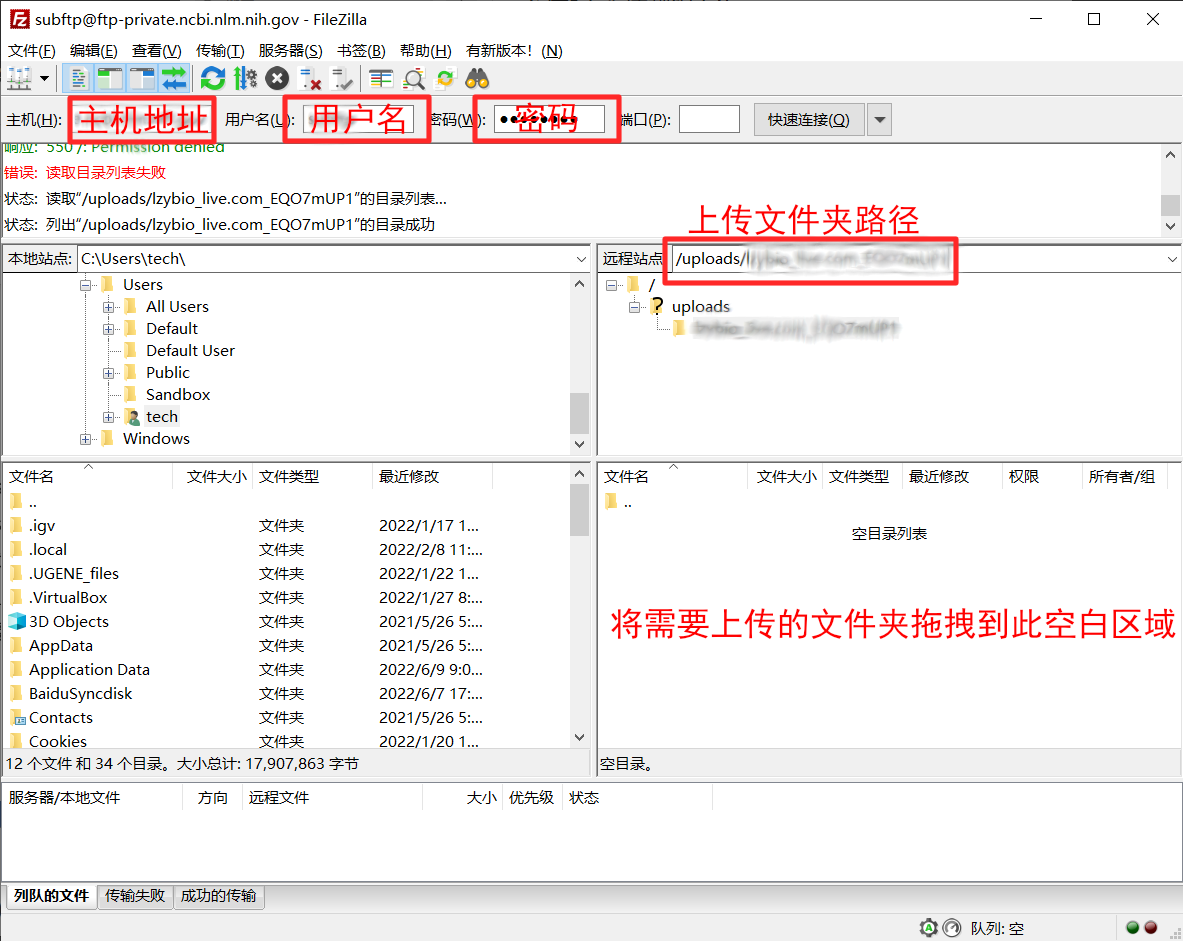
完全上传完成所有文件后,请您检查上传文件的大小是否与您电脑本地的同名文件相同,如果不同,请删除后重新上传。随后,请您回到 SRA 网页,点击“Select preload folder”按钮,然后在弹出界面中点击“Refresh folders”(上传完成后可能会需要等待十分钟才能刷新出来),点选您通过 FTP 上传的文件夹后,再点击“Use selected folder”。
图片
第八步:检查(Overview)
SRA 网页将会引导您**检查一遍上传的文件完整无误,随后点击“Submit”按钮。您需要等待 SRA 人工审核,审核通过后,您将会收到一封邮件通知,其中包括您这个项目的 SRA 登录号(SRA accession)。待数据公开后,其它研究者将可以通过该号码查询并下载您所上传的数据。
*温馨提示
由于 SRA 数据上传对于许多杂志的论文发表是必需的,而操作过程的繁琐以及网络的不稳定等因素又可能导致数据难以一次性上传成功,加之 SRA 需要多个工作日来检查您上传的数据,云序生物基于我们的经验,建议客户至少在截止日期一个月前开始进行 SRA 数据上传的工作,以免耽误您论文的正常发表。
